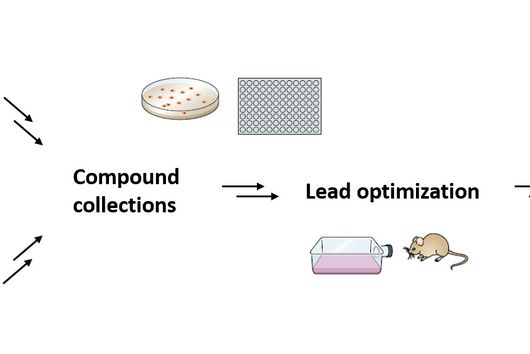
Saarbrücken, 11. September 2023 - Die Entdeckung von Arzneimitteln ist langwierig und mühsam, vor allem wenn es darum geht, Naturstoffe zu finden, die als Gerüst für potenzielle Arzneimittelkandidaten dienen könnten. Wie kann künstliche Intelligenz diesen Prozess beschleunigen? Ein Konsortium von Wissenschaftler:innen, darunter auch Prof. Olga Kalinina, Prof. Andrea Volkamer, Prof. Rolf Müller und Prof. Anna Hirsch vom Helmholtz-Institut für Pharmazeutische Forschung Saarland (HIPS), hat einen Übersichtsartikel über die aktuellen Anwendungen von künstlicher Intelligenz (KI) in der Naturstoffforschung verfasst und darüber, wie KI in Zukunft bei Projekten zur Wirkstofffindung helfen kann. Die Übersichtsarbeit ist ein direktes Ergebnis des " „Lorentz Workshop on AI and Natural Product Drug Discovery and Design“ ", der von Prof. Hirsch mitorganisiert wurde und im September 2021 stattfand. Das HIPS ist ein Standort des Helmholtz-Zentrums für Infektionsforschung (HZI) in Kooperation mit der Universität des Saarlandes.
Dank der Verfügbarkeit umfangreicher Omics-Daten und verbesserter Computerkapazitäten erlebt die Naturstoffforschung sowohl in der akademischen als auch in der industriellen Forschung eine Renaissance. Naturstoffe, auch Sekundärmetabolite genannt, sind eine vielversprechende Quelle für Wirkstoffe, da sie im Vergleich zu den meist „flachen" synthetischen Strukturen einen relativ hohen Grad an Dreidimensionalität aufweisen. Da diese Substanzen natürlichen Ursprungs sind, gibt es in der Regel Transportersysteme, die letztendlich dazu beitragen, dass der Wirkstoff sein Ziel erreicht. Die Vorhersage der molekularen Ziele der Wirkstoffkandidaten, ihrer biologischen Aktivitäten sowie möglicher Nebenwirkungen gehören zu den wichtigsten Anwendungsbereichen für KI in der Naturstoffforschung.
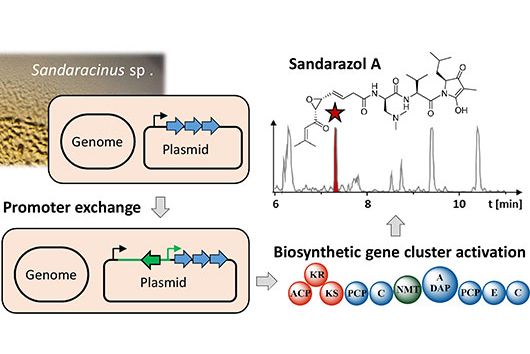
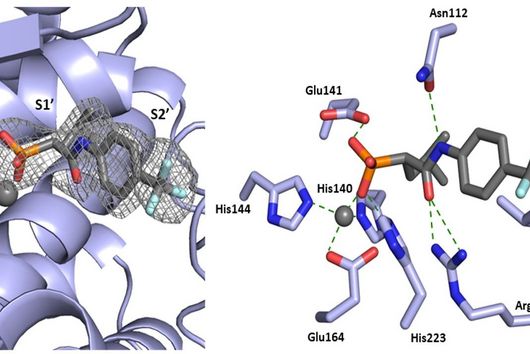
Derzeit konzentrieren sich KI-basierte Ansätze auf DNA-Sequenzen, um die chemischen Strukturen der Naturstoffe vorherzusagen, die von den biosynthetischen Genclustern (BGCs) produziert werden. Diese sind im Genom der produzierenden Organismen kodiert. Bislang wurden bereits über 2.500 BGCs und ihre Produkte experimentell charakterisiert. Dies ebnet den Weg für die computergestützte Genomanalyse, auch bekannt als Genom-Mining, um eine Vielzahl von Biosynthesewegen für neue Moleküle zu identifizieren. Eine zentrale Herausforderung für das Genom-Mining ist die Identifizierung neuartiger BGCs und nicht geclusterter Biosynthesewege, bei denen die Gensequenzen nicht in unmittelbarer Nähe zueinander, sondern mehr oder weniger verstreut über das Genom liegen. Gerade in diesen Bereichen können Algorithmen des maschinellen Lernens helfen, sie zu entdecken. Allerdings ist immer noch menschliche Expertise erforderlich, um die BGC-Grenzen als Eingabe für das maschinelle Lernen manuell zu aktualisieren. Außerdem müssen die Vorhersagemethoden für bioaktive Verbindungen weiter verbessert und erforscht werden.
In ihrem Übersichtsartikel beschreiben die Forschenden, dass der Mangel an qualitativ hochwertigen standardisierten Daten einer der Engpässe aktueller KI-Technologien ist, um ihr volles Potenzial auszuschöpfen. Diese Algorithmen sind im Allgemeinen auf Trainingsdatensätze angewiesen, die ausreichend sein sollten, um die Komplexität des Modells zu unterstützen. Erst dann kann ihre Leistung verbessert werden. Es ist von entscheidender Bedeutung, dass verschiedene Datensätze und Annotationsmethoden einheitlich sind, um beispielsweise die Entdeckung von Naturstoffen voranzutreiben. Die Autor:innen ermutigen die wissenschaftliche Gemeinschaft, eine standardisierte maschinenlesbare Datei für alle Verbindungen, die sie in ihren Veröffentlichungen beschreiben, zu erstellen. Diese Datei sollte die notwendigen Informationen wie die annotierte chemische Struktur, den Namen der Verbindung, den produzierenden Organismus und die BGC enthalten. Anschließend können diese Verbindungen in Datenbanken von Naturstoff-zentrierten Ressourcen importiert werden.
„KI-basierte Ansätze bieten die Chance, den langwierigen und teuren Prozess der Medikamentenentwicklung deutlich effizienter zu gestalten und somit schneller zu wirksamen und sicheren Medikamenten zu gelangen", sagt Anna Hirsch, eine der Korrespondenzautor:innen des Übersichtsartikels und Leiterin der Abteilung Wirkstoffdesign und Optimierung am HIPS. Außerdem seien die kollektiven Ressourcen der weltweiten wissenschaftlichen Community bei weitem größer als die Kapazitäten eines einzelnen Labors. Wenn allgemeine Leitlinien für die Erstellung von gemeinschaftlichen und kuratierten Datensätzen zur Verfügung stünden, könnte dies die Anwendung von KI bei der Entdeckung von Naturstoff-Arzneimitteln erheblich voranbringen.
Originalpublikation:
Marnix H. Medema und Kolleg:innen. Artificial intelligence for natural product drug discovery. Nature Reviews Drug Discovery, 2023. DOI: 10.1038/s41573-023-00774-7

![[Translate to German:] Künstliche Intelligenz unterstützt die Entdeckung von Arzneimitteln aus Naturstoffen](/fileadmin/HIPS/__processed__/3/e/csm_AI_in_NP_Drug_Discovery_AdobeStock_776f8854f2.jpg)
![[Translate to German:] Künstliche Intelligenz unterstützt die Entdeckung von Arzneimitteln aus Naturstoffen](/fileadmin/HIPS/__processed__/3/e/csm_AI_in_NP_Drug_Discovery_AdobeStock_cb35048970.jpg)









![[Translate to German:] [Translate to German:]](/fileadmin/HIPS/__processed__/c/9/csm_HZI_wiss_hemmhof_mik_001_611abc3df4.jpg)


![[Translate to German:] [Translate to German:]](/fileadmin/HIPS/__processed__/1/b/csm_Andreas_Keller_2023_org_4a279f0c07.jpg)
![[Translate to German:] [Translate to German:]](/fileadmin/HIPS/__processed__/b/3/csm_AufDiePlaetze_WID-8615_4ee837e55b.jpg)














![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIPS/__processed__/f/b/csm_Hegemann_Julian_a8a2ed3682.jpg)


![[Translate to Deutsch:] [Translate to Deutsch:]](/fileadmin/HIPS/__processed__/2/b/csm_HZI_wiss_pseudomonas_cpi_hzi_001_62730ae12d.jpg)