Wirkstoffdesign und Optimierung
Prof. Dr. Anna K. H. Hirsch
Die Hirsch-Gruppe verfolgt eine strukturbasierte rationale Designstrategie, bei der sie sich auf biologisch relevante, häufig wenig erforschte Enzyme, Transporter und Regulatoren aus Bakterien oder Parasiten konzentriert. Die Gruppe verwendet diverse biophysikalische Methoden, um Wechselwirkungen zwischen Substanzen und ihren Zielproteinen zu untersuchen, und setzt zahlreiche in vitro- und zellbasierte Assays zur Evaluierung neuartiger Anti-Infektiva ein. Basierend auf diesen Ergebnissen erfolgt die Multiparameter-Optimierung der Wirkstoffe.
Unsere Forschung
Das Auftreten Antibiotika-resistenter Stämme humanpathogener Bakterien wie P. aeruginosa, S. aureus oder S. pneumoniae ist eine nicht zu unterschätzende Bedrohung für die menschliche Gesundheit. Dies erfordert dringend die Entwicklung neuer antibakterieller Wirkstoffe mit neuen Wirkungsweisen. Die Abteilung von Prof. Anna Hirsch verfolgt eine strukturbasierte Strategie, die sich auf ein Portfolio zweier Arten von Wirkstofftargets konzentriert.
Die erste Gruppe umfasst Targets, die wichtige Mechanismen innerhalb des Bakteriums beeinträchtigen und diese dadurch effektiv abtöten. Ein Beispiel ist das Enzym DXS, das eine entscheidende Rolle im Methylerythritol-Phosphatweg spielt, der für die Biosynthese universeller isoprenoider Vorläufer in vielen gramnegativen Krankheitserregern unerlässlich ist, aber beim Menschen fehlt. Die zweite Gruppe umfasst Targets, die die Pathogenität und Virulenz der Bakterien beeinträchtigen, ohne aber deren Lebensfähigkeit herabzusetzen. Es wird angenommen, dass diese sogenannten Pathoblocker mit einer geringeren Wahrscheinlichkeit die Entwicklung von Resistenzen verursachen, während die Darmflora weitgehend unberührt bleibt.
Die Hirsch-Gruppe wendet eine Reihe etablierter Hit-Identifikationsstrategien an, darunter struktur- und fragmentbasiertes Wirkstoffdesign, klassische medizinische Chemie und virtuelles Screening. Darüber hinaus werden durch die Pionierarbeit bei innovativen Methoden wie der dynamischen kombinatorischen Chemie und der kinetischen zielgerichteten Synthese wichtige Engpässe, wie das Spektrum chemischer Reaktionen und biologischer Targets, angegangen. Der Einsatz etablierter und innovativer Techniken zur Entwicklung, Synthese und Profilierung der vielversprechendsten Inhibitoren ermöglicht eine effiziente anschließende Multiparameter-Optimierung sowie die Aufklärung der Wirkungsweise dieser Verbindungen.
Die Wissenschaftler des interdisziplinären Teams haben unterschiedliche Ausbildungs-Hintergründe. Hierzu zählen nicht nur die medizinische und synthetische organische Chemie, sondern auch die Pharmazie, Pharmakologie, Biologie und Biochemie, was zu einem vielfältigen Kompetenzspektrum führt.
Team-Mitglieder

Prof. Dr. Anna K. H. Hirsch
Gruppenleiterin

Bahareh Kadkhodazadeh
Assistentin

Nicole Klein Ramos
Assistentin

Dr. Godfrey Mayoka
Wissenschaftler

Dr. Jörg Haupenthal
Wissenschaftler

Dr. Andreas Kany
Postdoc

Dr. Christian Schütz
Postdoc

Dr. Danica Walsh
Postdoc

Dr. Gwenaëlle Jézéquel
Postdoc

Dr. Mariia Nesterkina
Postdoc

Dr. Mostafa Hamed
Postdoc

Dr. Walid A. M. Elgaher
Postdoc

Dr. Života Selaković
Postdoc

Ahmed Amin
Doktorand

Andreas Klein
Doktorand

Angeliki Vgenopoulou
Doktorandin

Antoine Lacour
Doktorand

Atanaz Shams
Doktorandin

Daan Willocx
Doktorand

Dominik Kolling
Doktorand

Gabriele Bianchi
Doktorand

Hugo Scherer
Doktorand

Ioulia Antonia Exapicheidou
Doktorandin

Justine Bassil
Doktorandin

Márió Széles
Doktorand

Roya Shafiei
Doktorandin

Sidra Eisa
Doktorandin

Jannine Seelbach
Technische Assistentin

Jeannine Jung
Technische Assistentin

Lizana Martínez
Technische Assistentin

Simone Amann
Technische Assistentin

Tabea Wittmann
Technische Assistentin
Forschungsprojekte
Da die Zahl der gegen gängige Antibiotika resistenten pathogenen Bakterien rapide zunimmt, ist die Entwicklung neuer antibakterieller Wirkstoffe dringend erforderlich. Um diese Probleme anzugehen, verfolgt die Hirsch-Gruppe eine Strategie, die auf dem rationalen Design und der Synthese von Antiinfektiva mit neuen Wirkungsweisen basiert. In diesem Zusammenhang konzentriert sich die Gruppe auf ein Portfolio von biologisch relevanten, idealerweise wenig erforschten Enzymen, Transportern und Regulatoren in bakteriellen Pathogenen. Diese Wirkstofftargets können in diejenigen gruppiert werden, die lebensnotwendige Mechanismen innerhalb des Bakteriums beeinträchtigen und diese somit effektiv abtöten (z.B. ECF-T, DnaN und DXS) und in diejenigen, die die Pathogenität und Virulenz herabsetzen, ohne jedoch die bakterielle Lebensfähigkeit zu beeinträchtigen (z.B. ColH und LasB).
Die Gruppe verwendet eine Vielzahl biophysikalischer Methoden, um neue Hitverbindungen zu identifizieren und um Wechselwirkungen zwischen einzelnen Verbindungen und ihren Zielproteinen zu untersuchen. Verschiedene in vitro- und zellbasierte Assays, die Generierung von in silico-Daten und die Co-Kristallisation ausgewählter Verbindungen mit ihren Zielproteinen unterstützen die Evaluierung neuartiger Antiinfektiva und deren weitere Optimierung durch klassische und innovative Ansätze der medizinischen Chemie.
Nachfolgend finden Sie eine Übersicht über ausgewählte Targets, die wir derzeit bearbeiten und auf die wir die oben genannten Strategien anwenden:
ECF-T
Die Transporter des Energie-Kopplungsfaktors (ECF) sind eine Familie von Transmembranproteinen, die an der Aufnahme von Vitaminen durch eine Vielzahl von Bakterien beteiligt sind. Die Hemmung der Aktivität dieser Proteine könnte die Lebensfähigkeit von Organismen beeinträchtigen, die von der Vitaminaufnahme abhängig sind. Dazu gehören Pathogene wie Staphylococcus aureus, Streptococcus pneumoniae und Enterococcus faecium. Da diese Bakterien keine wichtigen Vitamine wie Folsäure, Pantothenat und Niacin de novo synthetisieren können, sind sie auf ECF-Transporter angewiesen, die die Vitamine aus der Umwelt in die Zelle schleusen. Aufgrund dieser zentralen Rolle im Stoffwechsel solcher Bakterien sind ECF-Transporter neuartige potenzielle antimikrobielle Targets. Vor kurzem haben wir die ersten Klassen selektiver antibakterieller Wirkstoffe identifiziert, die eine vielversprechende Hemmung der ECF-Transporter zeigen. Aktuell versuchen wir, diese Verbindungen weiterzuentwickeln.
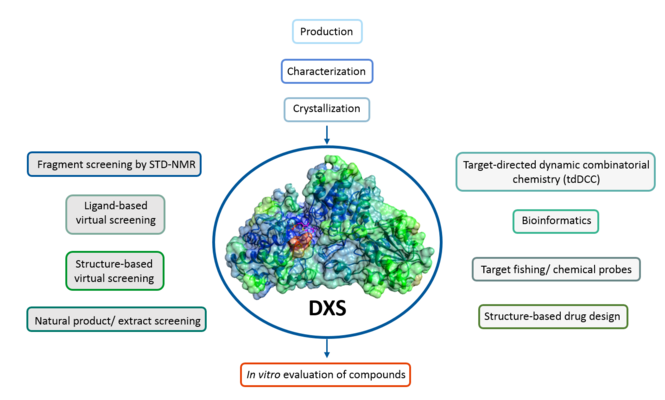
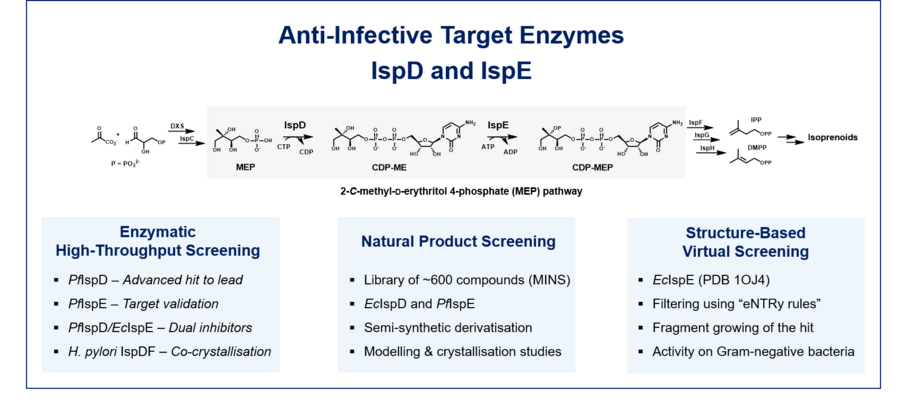
MEP-Signalweg (Targets: DXS, IspD, IspE)
Der Methylerythritolphosphat (MEP)-Signalweg ist eine reiche, wenig erforschte Quelle für antiinfektive Targets, die beim Menschen fehlen, aber in vielen pathogenen Bakterien, Protozoen und Pflanzen unerlässlich sind. Dies eröffnet die Möglichkeit, hochselektive Inhibitoren zu entwickeln, die gegen klinisch relevante Krankheitserreger wie beispielsweise vier ESKAPE-Erreger wirksam sind: Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa und Enterobacter spp., die laut der „WHO Global Priority List of antibiotic-resistant bacteria“ höchste Priorität haben - zuzüglich Mycobacterium tuberculosis und Plasmodium falciparum. Bei diesen Krankheitserregern ist der MEP-Signalweg die einzige Quelle für Isopentenyldiphosphat (IPP) und Dimethylallyldiphosphat (DMAPP). Letztere sind die universellen Bausteine für die Biosynthese von Isoprenoiden, einer großen und vielfältigen Klasse von Naturstoffen mit einer Vielzahl von unverzichtbaren Funktionen. Mit DXS, IspD und IspE sprechen wir drei essentielle Enzyme dieses Signalweges an. Unsere besten Verbindungen sind hochaktiv gegen die genannten Krankheitserreger und zeigen nanomolare IC50-Werte.
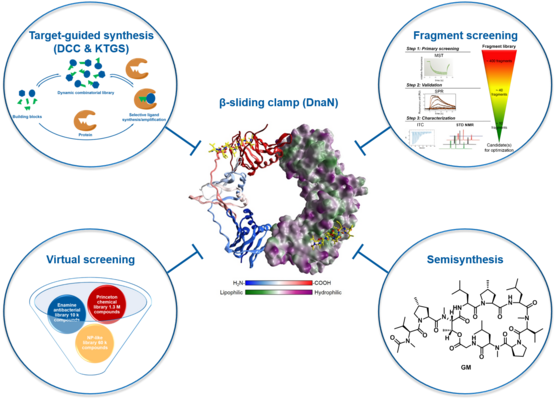
DnaN
Antibakterielle Medikamente, die auf essentielle Multi-Enzym-Komplexe abzielen, sind weniger anfällig für die Entwicklung von Resistenzen, da ihre Fehlfunktion schwer zu umgehen ist. In diesem Zusammenhang enthält die bakterielle Replizierungsmaschinerie, die aus mindestens zwölf interagierenden Enzymen besteht, die in Bakterien hochkonserviert sind, mehrere attraktive und vielversprechende antibakterielle Ziele - darunter die bakterielle β-Gleitklemme DnaN. DnaN ist eine wichtige Untereinheit des Holoenzyms DNA-Polymerase III, die die Polymerase-Dissoziation verhindert und die Polymerase-Aktivität erhöht. Darüber hinaus zeigt die β-Gleitklemme Wechselwirkungen mit einer Vielzahl anderer Enzyme, die an Replikations- und DNA-Reparaturprozessen beteiligt sind. Seine wichtige Funktionalität im Replikationsprozess und seine hochkonservierte Struktur über Bakterienarten hinweg sowie seine signifikante strukturelle Divergenz zum Säugetiergegenstück (PCNA) machen DnaN zu einem attraktiven Ziel für die Behandlung verschiedener Krankheitserreger, einschließlich Mycobacterium tuberculosis. Basierend auf dem Naturstoff Griselimycin (GM), einem bekannten DnaN-Binder, haben wir einen semisynthetischen Ansatz gewählt, der zu einer Reihe von GM-Derivaten mit vielversprechenden antibakteriellen Aktivitäten führte. Darüber hinaus haben wir verschiedene kleine organische Moleküle identifiziert, die an DnaN binden. Die Optimierung der vielversprechendsten Wirkstoffe ist im Gange.
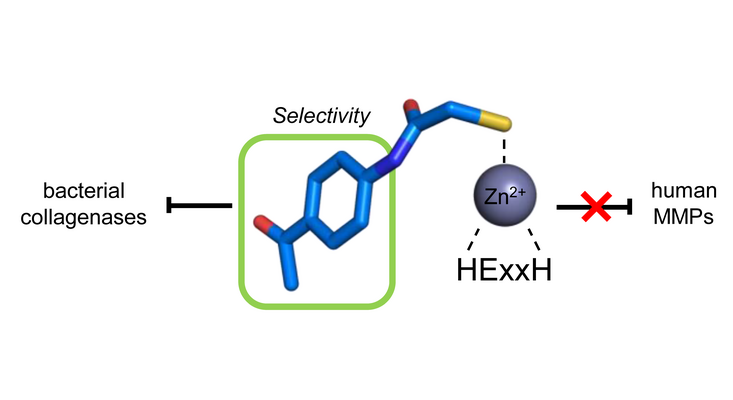
ColH
Clostridien stellen eine Familie von grampositiven Bakterien dar, die gefährliche Krankheitserreger umfassen, die schwere Infektionskrankheiten verursachen. Die hohe Letalität dieser Bakterien steht im Zusammenhang mit Kollagenasen, die für die clostridiale Virulenz entscheidend sind. Denn sie spielen eine entscheidende Rolle bei der Kolonisierung und Umgehung der Immunabwehr des Wirtes, dem Erwerb von Nährstoffen, der Erleichterung der Verbreitung oder der Gewebeschädigung während der Infektion. Die Hemmung dieser extrazellulären Kollagenasen ist konzeptionell attraktiv, da sie den Erreger nicht direkt angreift, sondern die Kolonisation und Infiltration des Wirtes durch die Clostridien blockiert. Die gezielte Anwendung extrazellulärer Enzyme hat den zusätzlichen Vorteil, dass Inhibitoren die Bakterienzellwand nicht durchqueren müssen, was in vielen Fällen eine Herausforderung darstellt. Clostridiale Kollagenasen sind Zink-Metalloproteinasen mit einer multidomänen Organisation, von denen Homologe auch in vielen Bazillen vorkommen. Unsere Gruppe war die erste, die Substanzen veröffentlichte, die nicht nur eine hochwirksame Hemmung der clostridialen und bazillalen Kollagenasen (ColH IC50 im nM-Bereich), sondern auch eine starke Selektivität gegenüber menschlichen MMPs zeigten.
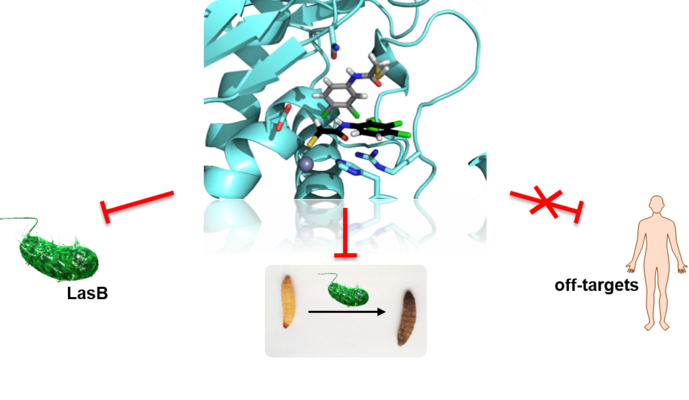
LasB
Pseudomonas aeruginosa (PA), ein äußerst problematischer gramnegativer Krankheitserreger, dem von der WHO eine kritische Priorität eingeräumt wurde, ist für viele nosokomiale Infektionen verantwortlich. Der opportunistische Erreger kommt auch häufig bei Verbrennungen und in der Lunge von COPD- und Mukoviszidosepatienten vor. Aus PA abgeleitete Virulenzfaktoren spielen eine zentrale Rolle bei Mechanismen, die ihre Pathogenese und Infektivität vermitteln. Es wird erwartet, dass die Strategie zur Entwicklung von "Pathoblockern", die auf solche Virulenzfaktoren abzielen, das kommensale Mikrobiom intakt lässt und weniger anfällig für die Entwicklung von Resistenzen ist als bei herkömmlichen Antibiotika. Einer der wichtigsten Virulenzfaktoren von PA ist die extrazelluläre Zink-Metall-Protease Elastase (LasB). LasB trägt wesentlich zum Krankheitsverlauf bei PA-infizierten Personen bei, indem es die Invasion des Wirts und die Umgehung der Immunabwehr erleichtert. Zudem wurde berichtet, dass es an der Bildung von PA-Biofilmen beteiligt ist. Die Hemmung von LasB ist eine vielversprechende Strategie zur effektiven Reduzierung der Virulenz von PA, ohne die Lebensfähigkeit des Erregers und des Wirtsmikrobioms zu beeinträchtigen. Unsere vielversprechendsten LasB-Inhibitoren weisen nanomolare IC50-Werte und ein vielversprechendes Sicherheitsprofil auf.
Publikationen
2024
High Target Homology Does Not Guarantee Inhibition: Aminothiazoles Emerge as Inhibitors of Plasmodium falciparum
Johannsen S, Gierse R, Krüger A, Edwards R, Nanna V, Fontana A, Di Zhu, Masini T, Carvalho L, Poizat M, …, Wrenger C, Hirsch A (2024)
ACS Infect. Dis.DOI: 10.1021/acsinfecdis.3c00670
Hit optimization by dynamic combinatorial chemistry on Streptococcus pneumoniae energy-coupling factor transporter ECF-PanT
Exapicheidou I, Shams A, Ibrahim H, Tsarenko A, Backenköhler M, Hamed M, Diamanti E, Volkamer A, slotboom d, Hirsch A (2024)
Chem. Commun. 60 (7): 870-873DOI: 10.1039/D3CC04738E
2023
Clean Synthetic Strategies to Biologically Active Molecules from Lignin: A Green Path to Drug Discovery
Afanasenko A, Wu X, Santi A, Elgaher W, Kany A, Shafiei R, Schulze M, Schulz T, Haupenthal J, Hirsch A, Barta K (2023)
Angew. Chem. Int. Ed.DOI: 10.1002/anie.202308131
Identification of HuR–RNA Interfering Compounds by Dynamic Combinatorial Chemistry and Fluorescence Polarization
Della Volpe S, Listro R, Ambrosio F, Garbagnoli M, Linciano P, Rossi D, Costa G, Alcaro S, Vasile F, Hirsch A, Collina S (2023)
ACS Med. Chem. Lett.DOI: 10.1021/acsmedchemlett.3c00303
1-deoxy-D-xylulose-5-phosphate synthase from Pseudomonas aeruginosa and Klebsiella pneumoniae reveals conformational changes upon cofactor binding
Hamid R, Adam S, Lacour A, Monjas L, Köhnke J, Hirsch A (2023)
The Journal of biological chemistryDOI: 10.1016/j.jbc.2023.105152
pH-Responsive Dynaplexes as Potent Apoptosis Inductors by Intracellular Delivery of Survivin siRNA
Liu Y, Ashmawy S, Latta L, Weiss A, Kiefer A, Nasr S, Loretz B, Hirsch A, Lee S, Lehr C (2023)
Biomacromolecules 24 (8): 3742-3754DOI: 10.1021/acs.biomac.3c00424
Exploring the Translational Gap of a Novel Class of Escherichia coli IspE Inhibitors
Ropponen H, Diamanti E, Johannsen S, Illarionov B, Hamid R, Jaki M, Sass P, Fischer M, Haupenthal J, Hirsch A (2023)
ChemMedChemDOI: 10.1002/cmdc.202300346
Inhibitors of the Elastase LasB for the treatment of Pseudomonas aeruginosa lung infections
Konstantinovic J, Kany A, Alhayek A, Abdelsamie A, Sikandar A, Voos K, Yao Y, Andreas A, Shafiei R, Loretz B, …, Haupenthal J, Hirsch A (2023)
Artificial intelligence for natural product drug discovery
Mullowney M, Duncan K, Elsayed S, Garg N, van der Hooft J, Martin N, Meijer D, Terlouw B, Biermann F, Blin K, …, Robinson S, Medema M (2023)
Nat Rev Drug DiscovDOI: 10.1038/s41573-023-00774-7
Thermoresponsive cholesteric liquid-crystal systems doped with terpenoids as drug delivery systems for skin applications
Nesterkina M, Vashchenko O, Vashchenko P, Lisetski L, Kravchenko I, Hirsch A, Lehr C (2023)
European journal of pharmaceutics and biopharmaceutics : official journal of Arbeitsgemeinschaft fur Pharmazeutische Verfahrenstechnik e.VDOI: 10.1016/j.ejpb.2023.09.002
Design of thiamine analogues for inhibition of thiamine diphosphate (ThDP)-dependent enzymes: Systematic investigation through Scaffold-Hopping and C2-Functionalisation
Chan A, Ho T, Irfan R, Hamid R, Rudge E, Iqbal A, Turner A, Hirsch A., Leeper F (2023)
Bioorganic chemistry 138DOI: 10.1016/j.bioorg.2023.106602
Facile Production of the Pseudomonas aeruginosa Virulence Factor LasB in E. coli for Structure-Based Drug Design
Kolling D, Haupenthal J, Hirsch A, Köhnke J (2023)
ChemBioChemDOI: 10.1002/cbic.202300185
Structure of Mycobacterium tuberculosis 1-Deoxy-D-Xylulose 5-Phosphate Synthase in Complex with Butylacetylphosphonate
Gawriljuk V, Oerlemans R, Gierse R, Jotwani R, Hirsch A, Groves M (2023)
Crystals 13 (5)DOI: 10.3390/cryst13050737
Not Every Hit-Identification Technique Works on 1-Deoxy-d-Xylulose 5-Phosphate Synthase (DXPS): Making the Most of a Virtual Screening Campaign
Johannsen S, Gierse R, Olshanova A, Smerznak E, Laggner C, Eschweiler L, Adeli Z, Hamid R, Alhayek A, Reiling N, Haupenthal J, Hirsch A (2023)
ChemMedChemDOI: 10.1002/cmdc.202200590
Genotoxic and mutational potential of monocyclic terpenoids (carvacrol, carvone and thymol) in Drosophila melanogaster
Nesterkina M, Bilokon S, Alieksieieva T, Kravchenko I, Hirsch A (2023)
Toxicol. Rep. 10DOI: 10.1016/j.toxrep.2023.02.009
Fighting antibiotic resistance-strategies and (pre)clinical developments to find new antibacterials
Walesch S, Birkelbach J, Jézéquel G, Haeckl F, Hegemann J, Hesterkamp T, Hirsch A, Hammann P, Müller R (2023)
EMBO reports 24 (1)DOI: 10.15252/embr.202256033
Towards Translation of PqsR Inverse Agonists: From In Vitro Efficacy Optimization to In Vivo Proof-of-Principle
Hamed M, Abdelsamie A, Rox K, Schütz C, Kany A, Röhrig T, Schmelz S, Blankenfeldt W, Arce-Rodriguez A, Borrero-de Acuña J, …, Hartmann R, Empting M (2023)
Adv. Sci. (Online)DOI: 10.1002/advs.202204443
2022
Discovery of the First Selective Nanomolar Inhibitors of ERAP2 by Kinetic Target‐Guided Synthesis
Camberlein V, Fléau C, Sierocki P, Li L, Gealageas R, Bosc D, Guillaume V, Warenghem S, Leroux F, Rosell M, …, Bouvier M, Deprez-Poulain R (2022)
Angew. Chemie 134 (39)DOI: 10.1002/ange.202203560
Synthesis, biological evaluation, and molecular docking studies of aldotetronic acid-based LpxC inhibitors
Wimmer S, Hoff K, Martin B, Grewer M, Denni L, Lascorz Massanet R, Raimondi M, Bülbül E, Melesina J, Hotop S, …, Sippl W, Holl R (2022)
Bioorganic chemistry 131DOI: 10.1016/j.bioorg.2022.106331
Proteoid biodynamers for safe mRNA transfection via pH-responsive nanorods enabling endosomal escape
Lee S, Nasr S, Rasheed S, Liu Y, Hartwig O, Kaya C, Boese A, Koch M, Herrmann J, Müller R, …, Hirsch A, Lehr C (2022)
Journal of controlled release : official journal of the Controlled Release Society 353: 915-929DOI: 10.1016/j.jconrel.2022.12.018
Baicalin lipid nanocapsules for treatment of glioma: characterization, mechanistic cytotoxicity, and pharmacokinetic evaluation
Ibrahim A, Abdel Gaber S, Fawzi Kabil M, Ahmed-Farid O, Hirsch A, El-Sherbiny I, Nasr M (2022)
Expert Opin. Drug Deliv. 19 (11): 1549-1560DOI: 10.1080/17425247.2022.2139370
Introduction to themed collection on fragment-based drug discovery
Rees D, Hirsch A, Erlanson D (2022)
RSC Med. Chem. 13DOI: 10.1039/D2MD90037H
An Efficient Way to Screen Inhibitors of Energy-Coupling Factor (ECF) Transporters in a Bacterial Uptake Assay
Bousis S, Winkler S, Haupenthal J, Fulco F, Diamanti E, Hirsch A (2022)
International journal of molecular sciences 23 (5)DOI: 10.3390/ijms23052637
Respiratory Syncytial Virus Two-Step Infection Screen Reveals Inhibitors of Early and Late Life Cycle Stages
Sake S, Kosch C, Blockus S, Haid S, Gunesch A, Zhang X, Friesland M, Trummer S, Grethe C, Kühnel A, …, Schulz T, Pietschmann T (2022)
Antimicrobial agents and chemotherapyDOI: 10.1128/aac.01032-22
Discovery and Characterization of Synthesized and FDA-Approved Inhibitors of Clostridial and Bacillary Collagenases
Alhayek A, Abdelsamie A, Schönauer E, Camberlein V, Hutterer E, Posselt G, Serwanja J, Blöchl C, Huber C, Haupenthal J, …, Wessler S, Hirsch A (2022)
Journal of medicinal chemistry 65 (19): 12933-12955DOI: 10.1021/acs.jmedchem.2c00785
Discovery of novel drug-like antitubercular hits targeting the MEP pathway enzyme DXPS by strategic application of ligand-based virtual screening
Di Zhu, Johannsen S, Masini T, Simonin C, Haupenthal J, Illarionov B, Andreas A, Awale M, Gierse R, van der Laan T, …, Reymond J, Hirsch A (2022)
Chem. Sci. 13 (36): 10686-10698DOI: 10.1039/D2SC02371G
Screening of Natural Products and Small Molecules Uncovers Novel Coronavirus 1a/1b Frameshifting Inhibitors with Antiviral Properties
Kibe A, Elgaher W, Rand U, Zimmer M, Kany A, Hermann J, Müller R, Cicin-Sain L, Hirsch A, Caliskan N (2022)
SSRN JournalDOI: 10.2139/ssrn.4157446
Design, Synthesis, Antimicrobial Activity, and Molecular Docking of Some New Diclofenac Derivatives
Tolba M, Hamed M, Sayed M, Kamal El-Dean A, Abdel-Mohsen S, Ibrahim O, Elgaher W, Hirsch A, Saddik A (2022)
Polycyclic Aromatic Compounds: 1-16DOI: 10.1080/10406638.2022.2102661
Structural analysis of 1-deoxy-D-xylulose 5-phosphate synthase from Pseudomonas aeruginosa and Klebsiella pneumoniae reveals conformational changes upon cofactor binding
Hamid R, Adam S, Lacour A, Gomez L, Hirsch A (2022)
bioRxivDOI: 10.1101/2022.07.04.498669
Identification of RAD51-BRCA2 Inhibitors Using N-Acylhydrazone-Based Dynamic Combinatorial Chemistry
Bagnolini G, Balboni B, Schipani F, Gioia D, Veronesi M, Franco F, Kaya C, Jumde R, Ortega J, Girotto S, …, Roberti M, Cavalli A (2022)
ACS Med. Chem. Lett. 13 (8): 1262-1269DOI: 10.1021/acsmedchemlett.2c00063
The Structures and Binding Modes of Small-Molecule Inhibitors of Pseudomonas aeruginosa Elastase LasB
Camberlein V, Jézéquel G, Haupenthal J, Hirsch A (2022)
Antibiotics 11 (8)DOI: 10.3390/antibiotics11081060
Transferring Microclusters of P. aeruginosa Biofilms to the Air-Liquid Interface of Bronchial Epithelial Cells for Repeated Deposition of Aerosolized Tobramycin
Horstmann J, Laric A, Boese A, Yildiz D, Röhrig T, Empting M, Frank N, Krug D, Müller R, Schneider-Daum N, Souza Carvalho-Wodarz C, Lehr C (2022)
ACS infectious diseases 8 (1): 137-149DOI: 10.1021/acsinfecdis.1c00444
Structure-Guided Optimization of Small-Molecule Folate Uptake Inhibitors Targeting the Energy-Coupling Factor Transporters
Kiefer A, Bousis S, Hamed M, Diamanti E, Haupenthal J, Hirsch A (2022)
J. Med. Chem.DOI: 10.1021/acs.jmedchem.1c02114
Citraconate inhibits ACOD1 (IRG1) catalysis, reduces interferon responses and oxidative stress, and modulates inflammation and cell metabolism
Chen F, Elgaher W, Winterhoff M, Büssow K, Waqas F, Graner E, Pires-Afonso Y, Casares Perez L, La Vega L, Sahini N, …, Hirsch A, Pessler F (2022)
Nature metabolism 4 (5): 534-546DOI: 10.1038/s42255-022-00577-x
Targeting Extracellular Bacterial Proteases for the Development of Novel Antivirulence Agents
Kaya C, Hirsch A (2022)
Chimia 76 (5)DOI: 10.2533/chimia.2022.402
Design and Synthesis of Novel Bis-Imidazolyl Phenyl Butadiyne Derivatives as HCV NS5A Inhibitors
Hamdy J, Emadeldin N, Hamed M, Frakolaki E, Katsamakas S, Vassilaki N, Zoidis G, Hirsch A, Abdel-Halim M, Abadi A (2022)
Pharmaceuticals 15 (5)DOI: 10.3390/ph15050632
First crystal structures of 1-deoxy-D-xylulose 5-phosphate synthase (DXPS) from Mycobacterium tuberculosis indicate a distinct mechanism of intermediate stabilization
Gierse R, Oerlemans R, Reddem E, Gawriljuk V, Alhayek A, Baitinger D, Jakobi H, Laber B, Lange G, Hirsch A, Groves M (2022)
Sci. Rep. 12 (1)DOI: 10.1038/s41598-022-11205-9
N-Aryl-2-iso-butylmercaptoacetamides: the discovery of highly potent and selective inhibitors of Pseudomonas aeruginosa virulence factor LasB and Clostridium histolyticum virulence factor ColH
Voos K, Yahiaoui S, Konstantinovic J, Schönauer E, Alhayek A, Sikandar A, Si Chaib K, Ramspoth T, Rox K, Haupenthal J, …, Ducho C, Hirsch A (2022)
ChemRxivDOI: 10.26434/chemrxiv-2022-fjrqr
Structure-Based Design of α-Substituted Mercaptoacetamides as Inhibitors of the Virulence Factor LasB from Pseudomonas aeruginosa
Kaya C, Walter I, Alhayek A, Shafiei R, Jézéquel G, Andreas A, Konstantinovic J, Schönauer E, Sikandar A, Haupenthal J, …, Hartmann R, Hirsch A (2022)
ACS Infect. Dis.DOI: 10.1021/acsinfecdis.1c00628
N-Aryl Mercaptopropionamides as Broad-Spectrum Inhibitors of Metallo-β-Lactamases
Kaya C, Konstantinovic J, Kany A, Andreas A, Kramer J, Brunst S, Weizel L, Rotter M, Frank D, Yahiaoui S, …, Wichelhaus T, Hirsch A (2022)
Journal of medicinal chemistry 65 (5): 3913-3922DOI: 10.1021/acs.jmedchem.1c01755
Metabolic profiling of S-praziquantel: Structure elucidation using the crystalline sponge method in combination with mass spectrometry and nuclear magnetic resonance
Rosenberger L, Jenniches J, Essen C, Khutia A, Kühn C, Marx A, Georgi K, Hirsch A, Hartmann R, Badolo L (2022)
Drug metabolism and disposition: the biological fate of chemicalsDOI: 10.1124/dmd.121.000663
Inhibition of Collagenase Q1 of Bacillus cereus as a Novel Antivirulence Strategy for the Treatment of Skin‐Wound Infections
Alhayek A, Khan E, Schönauer E, Däinghaus T, Shafiei R, Voos K, Han M, Ducho C, Posselt G, Wessler S, …, del Campo A, Hirsch A (2022)
Adv. Therap.DOI: 10.1002/adtp.202100222
Bacteriomimetic Liposomes Improve Antibiotic Activity of a Novel Energy-Coupling Factor Transporter Inhibitor
Drost M, Diamanti E, Fuhrmann K, Goes A, Shams A, Haupenthal J, Koch M, Hirsch A, Fuhrmann G (2022)
Pharmaceutics 14 (1)DOI: 10.3390/pharmaceutics14010004
Inhibitors of Pseudomonas aeruginosa virulence factor LasB
Ducho C, Hartmann R, Haupenthal J, Hirsch A, Kany A, Kaya C, Konstantinovic J, Voos K, Walter I, Yahiaoui S, …, Jumde R, Kiefer A (2022)
Patent A61K31/095; A61K31/10; A61K31/167; A61K31/4184; A61K31/426; A61K31/428; A61K31/4406; A61P31/04; C07C323/60; C07D213/74; C07D235/30; C07D277/46; C07D277/82; C07D333/36; C07F9/38; C07F9/40; (WO2022043322A1)
2021
Citatronic Acid and Derivates thereof for use as a medicament
Pessler F., Chen F., Winterhoff M., Büssow K., Blankenfeldt W., Hirsch A. K. H., Elgaher W (2021)
Patent (WO2022223778, 2022)
N-Phenyl-3-Mercaptopropanamide Derivatives as Metallo-Beta-Lactamase Inhibitors for the Treatment of Bacterial Infections
Hartmann R, Konstantinovic J, Haupenthal J, Hirsch A, Kany A, Kaya C, Yahiaoui S, Wichelhaus T, Proschak E (2021)
Patent A61K31/5375; A61P31/04; C07C233/15; C07C233/25; C07C233/33; C07C233/43; C07C233/54; C07C319/02; C07C323/52; C07D295/22; (WO2021191219A1)
New PqsR Inverse Agonist
Schütz C, Empting M, Ahmed S, Hamed M, Hartmann R, Röhrig T, Kany A, Hirsch A (2021)
Patent A61K31/4439; A61K31/506; A61P31/04; C07D401/12; C07D403/12; (WO2021136803A1)
Novel PqsR Inverse Agonists
Hamed M, Ahmed S, Empting. M., Schütz C, Hartmann R, Röhrig T, Kany A, Hirsch A (2021)
Patent A61K31/4439; A61P31/04; C07D401/14; C07D417/14; (WO2021136805A1)
Towards the sustainable discovery and development of new antibiotics
Miethke M, Pieroni M, Weber T, Brönstrup M, Hammann P, Halby L, Arimondo P, Glaser P, Aigle B, Bode H, …, Moser H, Müller R (2021)
Nature reviews. Chemistry 5 (10): 726-749DOI: 10.1038/s41570-021-00313-1
Targeting the IspD Enzyme in the MEP Pathway: Identification of a Novel Fragment Class
Diamanti E, Hamed M, Lacour A, Bravo P, Illarionov B, Fischer M, Rottmann M, Witschel M, Hirsch A (2021)
ChemMedChemDOI: 10.1002/cmdc.202100679
Substrate-Inspired Fragment Merging and Growing Affords Efficacious LasB Inhibitors
Kaya C, Walter I, Yahiaoui S, Sikandar A, Alhayek A, Konstantinovic J, Kany A, Haupenthal J, Köhnke J, Hartmann R, Hirsch A (2021)
Angewandte Chemie (International ed. in English)DOI: 10.1002/anie.202112295
Targeting the energy-coupling factor (ECF) transporters: identification of new tool compounds
Diamanti E, Setyawati I, Bousis S, Souza P, mojas l, Swier l, Haupenthal J, Gibson P, Volz C, stanek w, …, slotboom d, Hirsch A (2021)
ChemRxivDOI: 10.26434/chemrxiv-2021-xq08b-v2
Redesigning of the cap conformation and symmetry of the diphenylethyne core to yield highly potent pan-genotypic NS5A inhibitors with high potency and high resistance barrier
Abdallah M, Hamed M, Frakolaki E, Katsamakas S, Vassilaki N, Bartenschlager R, Zoidis G, Hirsch A, Abdel-Halim M, Abadi A (2021)
European journal of medicinal chemistryDOI: 10.1016/j.ejmech.2021.114034
Unveiling Adatoms in On-Surface Reactions: Combining Scanning Probe Microscopy with van't Hoff Plots
Moreno-López J, Pérez Paz A, Gottardi S, Solianyk L, Li J, Monjas L, Hirsch A, Mowbray D, Stöhr M (2021)
The journal of physical chemistry. C, Nanomaterials and interfaces 125 (18): 9847-9854DOI: 10.1021/acs.jpcc.1c03134
1st Spring Virtual Meeting on Medicinal Chemistry
Sousa M, Marques M, Faustino M (2021)
Chemistry Proceedings 4 (1)DOI: 10.3390/chemproc2021004001
Design, synthesis, and biological evaluation of novel benzimidazole derivatives as sphingosine kinase 1 inhibitor
Khairat S, Omar M, Ragab F, Roy S, Turab Naqvi A, Abdelsamie A, Hirsch A, Galal S, Hassan M, El Diwani H (2021)
Archiv der Pharmazie 354 (9)DOI: 10.1002/ardp.202100080
Identification of N,N-arylalkyl-picolinamide derivatives targeting the RNA-binding protein HuR, by combining biophysical fragment-screening and molecular hybridization
Della Volpe S, Linciano P, Listro R, Tumminelli E, Amadio M, Bonomo I, Elgaher W, Adam S, Hirsch A, Boeckler F, …, Rossi D, Collina S (2021)
Bioorganic chemistry 116DOI: 10.1016/j.bioorg.2021.105305
Expanding the Myxochelin Natural Product Family by Nicotinic Acid Containing Congeners
Frank N, Széles M, Akone S, Rasheed S, Hüttel S, Frewert S, Hamed M, Herrmann J, Schuler S, Hirsch A, Müller R (2021)
Molecules 26 (16)DOI: 10.3390/molecules26164929
N-Aryl mercaptoacetamides as potential multi-target inhibitors of metallo-β-lactamases (MBLs) and the virulence factor LasB from Pseudomonas aeruginosa
Yahiaoui S, Voos K, Haupenthal J, Wichelhaus T, Frank D, Weizel L, Rotter M, Brunst S, Kramer J, Proschak E, Ducho C, Hirsch A (2021)
RSC Med. Chem.DOI: 10.1039/D1MD00187F
Crystalline sponge affinity screening: A fast tool for soaking condition optimization without the need of X-ray diffraction analysis
Rosenberger L, Essen C, Khutia A, Kühn C, Georgi K, Hirsch A, Hartmann R, Badolo L (2021)
European journal of pharmaceutical sciences : official journal of the European Federation for Pharmaceutical Sciences 164DOI: 10.1016/j.ejps.2021.105884
A New PqsR Inverse Agonist Potentiates Tobramycin Efficacy to Eradicate Pseudomonas aeruginosa Biofilms
Schütz C, Ho D, Hamed M, Abdelsamie A, Röhrig T, Herr C, Kany A, Rox K, Schmelz S, Siebenbürger L, …, Lehr C, Empting M (2021)
Advanced science (Weinheim, Baden-Wurttemberg, Germany) 8 (12)DOI: 10.1002/advs.202004369
“Clicking“ fragment leads to novel dual-binding cholinesterase inhibitors
Moleda Z, Zawadzka A, Czarnocki Z, Monjas L, Hirsch A, Budzianowski A, Maurin J (2021)
Bioorg Med ChemDOI: 10.1016/j.bmc.2021.116269
Phosphonate as a Stable Zinc‐Binding Group for “Pathoblocker” Inhibitors of Clostridial Collagenase H (ColH) (ChemMedChem 8/2021)
Voos K, Schönauer E, Alhayek A, Haupenthal J, Andreas A, Müller R, Hartmann R, Brandstetter H, Hirsch A, Ducho C (2021)
ChemMedChem 16 (8): 1198-1198DOI: 10.1002/cmdc.202100229
Hit-optimization using target-directed dynamic combinatorial chemistry: Development of inhibitors of the anti-infective target 1-deoxy-D-xylulose-5-phosphate synthase
Jumde R, Guadigni M, Gierse R, Alhayek A, Di Zhu, Hamid Z, Johannsen S, Elgaher W, Neusens P, Nehls C, …, Reiling N, Hirsch A (2021)
Chemical ScienceDOI: 10.1039/D1SC00330E
Search for the Active Ingredients from a 2-Aminothiazole DMSO Stock Solution with Antimalarial Activity
Ropponen H, Bader C, Diamanti E, Illarionov B, Rottmann M, Fischer M, Witschel M, Müller R, Hirsch A (2021)
ChemMedChem 16 (13): 2089-2093DOI: 10.1002/cmdc.202100067
Mastering the Gram-Negative Bacterial Barrier - Chemical Approaches to Increase Bacterial Bioavailability of Antibiotics
Ropponen H, Richter R, Hirsch A, Lehr C (2021)
Advanced Drug Delivery ReviewsDOI: 10.1016/j.addr.2021.02.014
Assessment of the rules related to gaining activity against Gram-negative bacteria
Ropponen H, Diamanti E, Siemens A, Illarionov B, Haupenthal J, Fischer M, Rottmann M, Witschel M, Hirsch A (2021)
RSC Med. Chem.DOI: 10.1039/D0MD00409J
Enhancing glycan stability via site-selective fluorination: modulating substrate orientation by molecular design
Axer A, Jumde R, Adam S, Faust A, Schäfers M, Fobker M, Koehnke J, Hirsch A, Gilmour R (2021)
Chem. Sci. 12 (4): 1286-1294DOI: 10.1039/D0SC04297H
Identification of a 1-deoxy-D-xylulose-5-phosphate synthase (DXS) mutant with improved crystallographic properties
Gierse R, Reddem E, Alhayek A, Baitinger D, Hamid Z, Jakobi H, Laber B, Lange G, Hirsch A, Groves (2021)
Biochemical and Biophysical Research Communications 539DOI: 10.1016/j.bbrc.2020.12.069
2020
A hydrogel-based in vitro assay for the fast prediction of antibiotic accumulation in Gram-negative bacteria
Richter R, Kamal M, García-Rivera M, Kaspar J, Junk M, Elgaher W, Srikakulam S, Gress A, Beckmann A, Grißmer A, …, Schneider-Daum N, Lehr C (2020)
Materials today. Bio 8DOI: 10.1016/j.mtbio.2020.100084
Micro-rheological properties of lung homogenates correlate with infection severity in a mouse model of Pseudomonas aeruginosa lung infection
Murgia X, Kany A, Herr C, Ho D, Rossi C, Bals R, Lehr C, Hirsch A, Hartmann R, Empting M, Röhrig T (2020)
Scientific Reports 10 (1)DOI: 10.1038/s41598-020-73459-5
Semisynthesis and biological evaluation of amidochelocardin derivatives as broad-spectrum antibiotics
Grandclaudon C, Birudukota N, Elgaher W, Jumde R, Yahiaoui S, Arisetti N, Hennessen F, Hüttel S, Stadler M, Herrmann J, …, Hirsch A, Brönstrup M (2020)
European journal of medicinal chemistry 188DOI: 10.1016/j.ejmech.2019.112005
Discovery of Small-Molecule Stabilizers of 14-3-3 Protein-Protein Interactions via Dynamic Combinatorial Chemistry
Hartman A, Elgaher W, Hertrich N, Andrei S, Ottmann C, Hirsch A (2020)
ACS medicinal chemistry letters 11 (5): 1041-1046DOI: 10.1021/acsmedchemlett.9b00541
Evaluation of Bacterial RNA Polymerase Inhibitors in a Staphylococcus aureus-Based Wound Infection Model in SKH1 Mice
Haupenthal J, Kautz Y, Elgaher W, Pätzold L, Röhrig T, Laschke M, Tschernig T, Hirsch A, Molodtsov V, Murakami K, Hartmann R, Bischoff M (2020)
ACS infectious diseases 6 (10): 2573-2581DOI: 10.1021/acsinfecdis.0c00034
Protein‐Templated Hit Identification through an Ugi Four‐Component Reaction
Federica Mancini, M. Yagiz Unver, Walid A. M. Elgaher, Varsha R. Jumde, Alaa Alhayek, Peer Lukat, Jennifer Herrmann, Martin D. Witte, Matthias Köck, Wulf Blankenfeldt, Rolf Müller, Hirsch A (2020)
Chemistry – A European Journal 26 (64)DOI: 10.1002/chem.202086462
7-Hydroxycoumarins are Affinity-based Fluorescent Probes for Competitive Binding Studies of Macrophage Migration Inhibitory Factor
Xiao Z, Chen D, Song S, van der Vlag R, van der Wouden P, van Merkerk R, Cool R, Hirsch A, Melgert B, Quax W, Poelarends G, Dekker F (2020)
Journal of medicinal chemistryDOI: 10.1021/acs.jmedchem.0c01160
pH-Dependent morphology and optical properties of lysine-derived molecular biodynamers
Lee S, Kaya C, Jang H, Koch M, Loretz B, Buhler E, Lehr C, Hirsch A (2020)
Mater. Chem. Front. 4 (3): 905-909DOI: 10.1039/C9QM00651F
Rapid Discovery of Aspartyl Protease Inhibitors Using an Anchoring Approach
Konstantinidou M, Magari F, Sutanto F, Haupenthal J, Jumde R, Ünver M, Heine A, Camacho C, Hirsch A, Klebe G, Dömling A (2020)
ChemMedChem 15 (8): 680-684DOI: 10.1002/cmdc.202000024
Tracheal brush cells release acetylcholine in response to bitter tastants for paracrine and autocrine signaling
Hollenhorst M, Jurastow I, Nandigama R, Appenzeller S, Li L, Vogel J, Wiederhold S, Althaus M, Empting M, Altmüller J, …, Saliba A, Krasteva-Christ G (2020)
FASEB j. 34 (1): 316-332DOI: 10.1096/fj.201901314RR
Optimized Inhibitors of MDM2 via an Attempted Protein‐Templated Reductive Amination
Vlag R, Yagiz Unver M, Felicetti T, Twarda-Clapa A, Kassim F, Ermis C, Neochoritis C, Musielak B, Labuzek B, Dömling A, Holak T, Hirsch A (2020)
ChemMedChem 15 (4): 370-375DOI: 10.1002/cmdc.201900574
BOPC1 Enantiomers Preparation and HuR Interaction Study. From Molecular Modeling to a Curious DEEP-STD NMR Application
Della Volpe S, Listro R, Parafioriti M, Di Giacomo M, Rossi D, Ambrosio F, Costa G, Alcaro S, Ortuso F, Hirsch A, Vasile F, Collina S (2020)
ACS medicinal chemistry letters 11 (5): 883-888DOI: 10.1021/acsmedchemlett.9b00659
Protein‐Templated Hit Identification via an Ugi Four‐Component Reaction
Mancini F, Unver M, Elgaher W, Jumde V, Alhayek A, Lukat P, Herrmann J, Witte M, Köck M, Blankenfeldt W, Müller R, Hirsch A (2020)
Chem. Eur. J.DOI: 10.1002/chem.202002250
Validating the 1,2-Difluoro Motif As a Hybrid Bioisostere of CF3 and Et Using Matrix Metalloproteinases As Structural Probes
Erdeljac N, Thiehoff C, Jumde R, Daniliuc C, Höppner S, Faust A, Hirsch A, Gilmour R (2020)
Journal of medicinal chemistry 63 (11): 6225-6237DOI: 10.1021/acs.jmedchem.0c00648
Synthesis and Biological Evaluation of Novel 2-Substituted Analogues of (–)-Pentenomycin I
Zisopoulou S, Bousis S, Haupenthal J, Herrmann J, Müller R, Hirsch A, Komiotis D, Gallos J, Stathakis C (2020)
Synlett 31 (05): 475-481DOI: 10.1055/s-0039-1690772
N -Aryl-3-mercaptosuccinimides as Antivirulence Agents Targeting Pseudomonas aeruginosa Elastase and Clostridium Collagenases
Konstantinovic J, Yahiaoui S, Alhayek A, Haupenthal J, Schönauer E, Andreas A, Kany A, Müller R, Koehnke J, Berger F, …, Brandstetter H, Hirsch A (2020)
J. Med. Chem. 63 (15): 8359-8368DOI: 10.1021/acs.jmedchem.0c00584
Flotillin-mediated membrane fluidity controls peptidoglycan synthesis and MreB movement
Zielinska A, Savietto A, Sousa Borges A, Martinez D, Berbon M, Roelofsen J, Hartman A, Boer R, van der Klei I, Hirsch A, …, Bramkamp M, Scheffers D (2020)
eLife 9DOI: 10.7554/eLife.57179
Potential Dental Biofilm Inhibitors: Dynamic Combinatorial Chemistry Affords Sugar-Based Molecules that Target Bacterial Glucosyltransferase
Hartman A, Jumde V, Elgaher W, Te Poele E, Dijkhuizen L, Hirsch A (2020)
ChemMedChemDOI: 10.1002/cmdc.202000222
A rapid synthesis of low-nanomolar divalent LecA inhibitors in four linear steps from d -galactose pentaacetate
Zahorska E, Kuhaudomlarp S, Minervini S, Yousaf S, Lepsik M, Kinsinger T, Hirsch A, Imberty A, Titz A (2020)
Chem. Commun. 56 (62): 8822-8825DOI: 10.1039/d0cc03490h
Novel PqsR Inverse Agonists
Hamed M, Ahmed S, Empting. M., Schütz C, Hartmann R, Röhrig T, Kany A, Hirsch A (2020)
Patent (EP20150119)
2019
Energy-Coupling Factor Transporters as Novel Antimicrobial Targets
Bousis S, Setyawati I, Diamanti E, Slotboom D, Hirsch A (2019)
Advanced Therapeutics, 2, 1800066DOI: 10.1002/ADTP.201800066
Protein-Templated Dynamic Combinatorial Chemistry: Brief Overview and Experimental Protocol
Hartman A, Gierse R, Hirsch A (2019)
European journal of organic chemistry 2019 (22): 3581-3590DOI: 10.1002/ejoc.201900327
Concepts and Core Principles of Fragment-Based Drug Design
Kirsch P, Hartman A, Hirsch A, Empting M (2019)
Molecules (Basel, Switzerland) 24 (23)DOI: 10.3390/molecules24234309
Comparing the Self-Assembly of Sexiphenyl-Dicarbonitrile on Graphite and Graphene on Cu(111)
Schmidt N, Li J, Gottardi S, Moreno-Lopez J, Enache M, Monjas L, van der Vlag R, Havenith R, Hirsch A, Stöhr M (2019)
Chem. Eur. J. 25 (19): 5065-5070DOI: 10.1002/chem.201806312
Inverting Small Molecule–Protein Recognition by the Fluorine Gauche Effect: Selectivity Regulated by Multiple H→F Bioisosterism
Bentler P, Bergander K, Daniliuc C, Mück-Lichtenfeld C, Jumde R, Hirsch A, Gilmour R (2019)
Angewandte Chemie (International ed. in English) 58 (32): 10990-10994DOI: 10.1002/anie.201905452
Spray-drying of inhalable, multifunctional formulations for the treatment of biofilms formed in cystic fibrosis
Lababidi N, Ofosu Kissi E, Elgaher W, Sigal V, Haupenthal J, Schwarz B, Hirsch A, Rades T, Schneider M (2019)
Journal of Controlled Release 314: 62-71DOI: 10.1016/j.jconrel.2019.10.038
From Wood to Tetrahydro-2-benzazepines in Three Waste-Free Steps: Modular Synthesis of Biologically Active Lignin-Derived Scaffolds
Elangovan S, Afanasenko A, Haupenthal J, Sun Z, Liu Y, Hirsch A, Barta K (2019)
ACS Cent. Sci. 5 (10): 1707-1716DOI: 10.1021/acscentsci.9b00781
Surface state tunable energy and mass renormalization from homothetic quantum dot arrays
Piquero-Zulaica I, Li J, Abd El-Fattah Z, Solianyk L, Gallardo I, Monjas L, Hirsch A, Arnau A, Ortega J, Stöhr M, Lobo-Checa J (2019)
Nanoscale 11 (48): 23132-23138DOI: 10.1039/c9nr07365e
Novel Compounds Targeting the RNA-Binding Protein HuR. Structure-Based Design, Synthesis, and Interaction Studies
Della Volpe S, Nasti R, Queirolo M, Unver M, Jumde V, Dömling A, Vasile F, Potenza D, Ambrosio F, Costa G, …, Hirsch A, Collina S (2019)
ACS Med. Chem. Lett. 10 (4): 615-620DOI: 10.1021/acsmedchemlett.8b00600
A combinatorial approach for the discovery of drug-like inhibitors of 15-lipoxygenase-1
van der Vlag R, Guo H, Hapko U, Eleftheriadis N, Monjas L, Dekker F, Hirsch A (2019)
European journal of medicinal chemistry 174: 45-55DOI: 10.1016/j.ejmech.2019.04.021
Rational Adaptation of L3MBTL1 Inhibitors to Create Small-Molecule Cbx7 Antagonists
Simhadri C, Daze K, Douglas S, Milosevich N, Monjas L, Dev A, Brown T, Hirsch A, Wulff J, Hof F (2019)
ChemMedChem 14 (15): 1444-1456DOI: 10.1002/cmdc.201900021
Low-Dimensional Metal-Organic Coordination Structures on Graphene
Li J, Solianyk L, Schmidt N, Baker B, Gottardi S, Moreno Lopez J, Enache M, Monjas L, van der Vlag R, Havenith R, Hirsch A, Stöhr M (2019)
The journal of physical chemistry. C, Nanomaterials and interfaces 123 (20): 12730-12735DOI: 10.1021/acs.jpcc.9b00326
Novel 15-Lipoxygenase-1 Inhibitor Protects Macrophages from Lipopolysaccharide-Induced Cytotoxicity
Guo H, Verhoek I, Prins G, van der Vlag R, van der Wouden P, van Merkerk R, Quax W, Olinga P, Hirsch A, Dekker F (2019)
Journal of medicinal chemistry 62 (9): 4624-4637DOI: 10.1021/acs.jmedchem.9b00212
Replacement of an Indole Scaffold Targeting Human 15-Lipoxygenase-1 Using Combinatorial Chemistry
Prismawan D, van der Vlag R, Guo H, Dekker F, Hirsch A (2019)
HCA 102 (5)DOI: 10.1002/hlca.201900040
Reversible immobilization of a protein to a gold surface through multiple host-guest interactions
Schwarz D, Elgaher W, Hollemeyer K, Hirsch A, Wenz G (2019)
J. Mater. Chem. B 7 (40): 6148-6155DOI: 10.1039/c9tb00560a
2018
Trendbericht Biochemie 2017: Proteinvermittelte dynamische kombinatorische Chemie
Hirsch A (2018)
Nachr. Chem. 66 (3): 281-283DOI: 10.1002/nadc.20184071734
Druggability Assessment of Targets Used in Kinetic Target-Guided Synthesis
Unver M, Gierse R, Ritchie H, Hirsch A (2018)
Journal of medicinal chemistry 61 (21): 9395-9409DOI: 10.1021/acs.jmedchem.8b00266
Phage Display on the Anti-infective Target 1-Deoxy-d-xylulose-5-phosphate Synthase Leads to an Acceptor-Substrate Competitive Peptidic Inhibitor
Marcozzi A, Masini T, Di Zhu, Pesce D, Illarionov B, Fischer M, Herrmann A, Hirsch A (2018)
ChemBioChem 19 (1): 58-65DOI: 10.1002/cbic.201700402
Lipid-DNAs as Solubilizers of m THPC
Liu Y, de Vries J, Liu Q, Hartman A, Wieland G, Wieczorek S, Börner H, Wiehe A, Buhler E, Stuart M, …, Herrmann A, Hirsch A (2018)
Chem. Eur. J. 24 (4): 798-802DOI: 10.1002/chem.201705206
Glucansucrase (mutant) enzymes from Lactobacillus reuteri 180 efficiently transglucosylate Stevia component rebaudioside A, resulting in a superior taste
Te Poele E, Devlamynck T, Jäger M, Gerwig G, van de Walle D, Dewettinck K, Hirsch A, Kamerling J, Soetaert W, Dijkhuizen L (2018)
Sci Rep 8 (1)DOI: 10.1038/s41598-018-19622-5
Delivery system for budesonide based on lipid-DNA
Liu Y, Bos I, Oenema T, Meurs H, Maarsingh H, Hirsch A (2018)
European journal of pharmaceutics and biopharmaceutics : official journal of Arbeitsgemeinschaft fur Pharmazeutische Verfahrenstechnik e.V 130: 123-127DOI: 10.1016/j.ejpb.2018.06.012
Dynamic Proteoids Generated From Dipeptide-Based Monomers
Liu Y, Stuart M, Buhler E, Hirsch A (2018)
Macromolecular rapid communications 39 (13)DOI: 10.1002/marc.201800099
Design and Synthesis of Bioisosteres of Acylhydrazones as Stable Inhibitors of the Aspartic Protease Endothiapepsin
Jumde V, Mondal M, Gierse R, Unver M, Magari F, van Lier R, Heine A, Klebe G, Hirsch A (2018)
ChemMedChem 13 (21): 2266-2270DOI: 10.1002/cmdc.201800446
Exploration of ligand binding modes towards the identification of compounds targeting HuR: a combined STD-NMR and Molecular Modelling approach
Vasile F, Della Volpe S, Ambrosio F, Costa G, Unver M, Zucal C, Rossi D, Martino E, Provenzani A, Hirsch A, …, Potenza D, Collina S (2018)
Sci Rep 8 (1)DOI: 10.1038/s41598-018-32084-z
Donepezil-melatonin hybrids as butyrylcholinesterase inhibitors: Improving binding affinity through varying mode of linking fragments
Lozinska I, Swierczynska A, Moleda Z, Hartman A, Hirsch A, Czarnocki Z (2018)
Archiv der Pharmazie 351 (11)DOI: 10.1002/ardp.201800194
2017
Molecular Biodynamers: Dynamic Covalent Analogues of Biopolymers
Liu Y, Lehn J, Hirsch A (2017)
Accounts of chemical research 50 (2): 376-386DOI: 10.1021/acs.accounts.6b00594
DXS as a target for structure-based drug design
Gierse R, Redeem E, Diamanti E, Wrenger C, Groves M, Hirsch A (2017)
Future medicinal chemistry 9 (11): 1277-1294DOI: 10.4155/fmc-2016-0239
Molecular insight into specific 14-3-3 modulators: Inhibitors and stabilisers of protein-protein interactions of 14-3-3
Hartman A, Hirsch A (2017)
European journal of medicinal chemistry 136DOI: 10.1016/j.ejmech.2017.04.058
Compounds Interfering with Embryonic Lethal Abnormal Vision (ELAV) Protein-RNA Complexes: An Avenue for Discovering New Drugs
Nasti R, Rossi D, Amadio M, Pascale A, Unver M, Hirsch A, Collina S (2017)
Journal of medicinal chemistry 60 (20): 8257-8267DOI: 10.1021/acs.jmedchem.6b01871
Fine-tuning Nanocarriers Specifically toward Cargo: A Competitive Study on Solubilizing Related Photosensitizers for Photodynamic Therapy
Wieczorek S, Remmler D, Masini T, Kochovski Z, Hirsch A, Börner H (2017)
Bioconjugate chemistry 28 (3): 760-767DOI: 10.1021/acs.bioconjchem.6b00549
Pentapeptide‐rich peptidoglycan at the Bacillus subtilis cell‐division site
Morales Angeles D, Liu Y, Hartman A, Borisova M, Sousa Borges A, Kok N, Beilharz K, veening j, Mayer C, Hirsch A, Scheffers D (2017)
Molecular microbiology 104 (2): 319-333DOI: 10.1111/mmi.13629
Bicyclic enol cyclocarbamates inhibit penicillin-binding proteins
Dockerty P, Edens J, Tol M, Morales Angeles D, Domenech A, Liu Y, Hirsch A, veening j, Scheffers D, Witte M (2017)
Organic & biomolecular chemistry 15 (4): 894-910DOI: 10.1039/c6ob01664b
Designed Spiroketal Protein Modulation
Scheepstra M, Andrei S, Unver M, Hirsch A, Leysen S, Ottmann C, Brunsveld L, Milroy L (2017)
Angewandte Chemie (International ed. in English) 56 (20): 5480-5484DOI: 10.1002/anie.201612504
Insight into the complete substrate-binding pocket of ThiT by chemical and genetic mutations
Swier L, Monjas L, Reeßing F, Oudshoorn R, Aisyah, Primke T, Bakker M, van Olst E, Ritschel T, Faustino I, …, Hirsch A, Slotboom D (2017)
MedChemComm 8 (5): 1121-1130DOI: 10.1039/c7md00079k
Saccharide-Containing Dynamic Proteoids
Liu Y, Stuart M, Witte M, Buhler E, Hirsch A (2017)
Chemistry (Weinheim an der Bergstrasse, Germany) 23 (64): 16162-16166DOI: 10.1002/chem.201703584
Dynamic Combinatorial Chemistry to Identify Binders of ThiT, an S-Component of the Energy-Coupling Factor Transporter for Thiamine
Monjas L, Swier L, Setyawati I, slotboom d, Hirsch A (2017)
ChemMedChem 12 (20): 1693-1696DOI: 10.1002/cmdc.201700440
2016
Design and synthesis of thiamine analogues to study their binding to the ECF transporter for thiamine in bacteria
Monjas L, Swier L, Voogd A, Oudshoorn R, Hirsch A, Slotboom D (2016)
Med. Chem. Commun. 7 (5): 966-971DOI: 10.1039/C6MD00022C
Furoates and thenoates inhibit pyruvate dehydrogenase kinase 2 allosterically by binding to its pyruvate regulatory site
Masini T, Birkaya B, van Dijk S, Mondal M, Hekelaar J, Jäger M, van Terwisscha Scheltinga A, Patel M, Hirsch A, Moman E (2016)
Journal of enzyme inhibition and medicinal chemistry 31 (sup4): 170-175DOI: 10.1080/14756366.2016.1201812
Proteoid Dynamers with Tunable Properties
Liu Y, Stuart M, Buhler E, Lehn J, Hirsch A (2016)
Fragment Linking and Optimization of Inhibitors of the Aspartic Protease Endothiapepsin: Fragment-Based Drug Design Facilitated by Dynamic Combinatorial Chemistry
Mondal M, Radeva N, Fanlo-Virgós H, Otto S, Klebe G, Hirsch A (2016)
Angewandte Chemie (International ed. in English) 55 (32): 9422-6DOI: 10.1002/anie.201603074
Fragment-Based Drug Design Facilitated by Protein-Templated Click Chemistry: Fragment Linking and Optimization of Inhibitors of the Aspartic Protease Endothiapepsin
Mondal M, Unver M, Pal A, Bakker M, Berrier S, Hirsch A (2016)
Chem. Eur. J. 22 (42): 14826-14830DOI: 10.1002/chem.201603001
2015
Dynamic combinatorial chemistry: a tool to facilitate the identification of inhibitors for protein targets
Mondal M, Hirsch A (2015)
Chemical Society reviews 44 (8): 2455-88DOI: 10.1039/c4cs00493k
Fighting malaria: structure-guided discovery of nonpeptidomimetic plasmepsin inhibitors
Huizing A, Mondal M, Hirsch A (2015)
Journal of medicinal chemistry 58 (13): 5151-63DOI: 10.1021/jm5014133
Harnessing dynamic combinatorial chemistry in the search for new ligands for protein targets
Monjas L, Hirsch A (2015)
Future medicinal chemistry 7 (16): 2095-8DOI: 10.4155/fmc.15.146
Combinatorial screening for specific drug solubilizers with switchable release profiles
Wieczorek S, Vigne S, Masini T, Ponader D, Hartmann L, Hirsch A, Börner H (2015)
Macromolecular Bioscience 15 (1): 82-9DOI: 10.1002/mabi.201400443
Structure-based design of potent small-molecule binders to the S-component of the ECF transporter for thiamine
Swier L, Monjas L, Guskov A, Voogd A, Erkens G, slotboom d, Hirsch A (2015)
ChemBioChem 16 (5): 819-26DOI: 10.1002/cbic.201402673
Fragment growing exploiting dynamic combinatorial chemistry of inhibitors of the aspartic protease endothiapepsin
Mondal M, Groothuis D, Hirsch A (2015)
DOI: 10.1039/C5MD00157A
Structure-Based Optimization of Inhibitors of the Aspartic Protease Endothiapepsin
Hartman A, Mondal M, Radeva N, Klebe G, Hirsch A (2015)
International journal of molecular sciences 16 (8): 19184-94DOI: 10.3390/ijms160819184
Supramolecular chemistry … and beyond
Hirsch A (2015)
Angewandte Chemie (International ed. in English) 54 (38): 11013-4DOI: 10.1002/anie.201506536
Validation of a homology model of Mycobacterium tuberculosis DXS: rationalization of observed activities of thiamine derivatives as potent inhibitors of two orthologues of DXS
Masini T, Lacy B, Monjas L, Hawksley D, Voogd A, Illarionov B, Iqbal A, Leeper F, Fischer M, Kontoyianni M, Hirsch A (2015)
Organic & biomolecular chemistry 13 (46): 11263-77DOI: 10.1039/c5ob01666e
Novel PqsR Inverse Agonists
Hamed M (2015)
Patent (EP 201523196)
2014
Development of inhibitors of the 2C-methyl-D-erythritol 4-phosphate (MEP) pathway enzymes as potential anti-infective agents
Masini T, Hirsch A (2014)
Journal of medicinal chemistry 57 (23): 9740-63DOI: 10.1021/jm5010978
Theoretical and structural analysis of long C-C bonds in the adducts of polycyanoethylene and anthracene derivatives and their connection to the reversibility of Diels-Alder reactions
Hirsch A, Reutenauer P, Le Moignan M, Ulrich S, Boul P, Harrowfield J, Jarowski P, Lehn J (2014)
Chemistry (Weinheim an der Bergstrasse, Germany) 20 (4): 1073-80DOI: 10.1002/chem.201303276
Structure-based design of inhibitors of the aspartic protease endothiapepsin by exploiting dynamic combinatorial chemistry
Mondal M, Radeva N, Köster H, Park A, Potamitis C, Zervou M, Klebe G, Hirsch A (2014)
Angewandte Chemie (International ed. in English) 53 (12): 3259-63DOI: 10.1002/anie.201309682
A natural-product switch for a dynamic protein interface
Scheepstra M, Nieto L, Hirsch A, Fuchs S, Leysen S, Lam C, het Panhuis L, van Boeckel C, Wienk H, Boelens R, …, Milroy L, Brunsveld L (2014)
Angewandte Chemie (International ed. in English) 53 (25): 6443-8DOI: 10.1002/anie.201403773
A doubly hermaphroditic chiral crown ether
Hirsch A, Sirlin C, Harrowfield J, Lehn J (2014)
DOI: 10.1039/C4CE00879K
De novo fragment-based design of inhibitors of DXS guided by spin-diffusion-based NMR spectroscopy
Masini T, Pilger J, Kroezen B, Illarionov B, Lottmann P, Fischer M, Griesinger C, Hirsch A (2014)
Chem. Sci. 5 (9): 3543-3551DOI: 10.1039/C4SC00588K
2013
Druggability of the enzymes of the non-mevalonate-pathway
Masini T, Kroezen B, Hirsch A (2013)
Drug Discovery Today 18 (23-24): 1256-62DOI: 10.1016/j.drudis.2013.07.003
Total synthesis, stereochemical elucidation and biological evaluation of Ac 2 SGL; a 1,3-methyl branched sulfoglycolipid from Mycobacterium tuberculosis
Geerdink D, Horst B, Lepore M, Mori L, Puzo G, Hirsch A, Gilleron M, Libero G, Minnaard A (2013)
DOI: 10.1039/C2SC21620E
Imidazole- and Benzimidazole-Based Inhibitors of the Kinase IspE: Targeting the Substrate-Binding Site and the Triphosphate-Binding Loop of the ATP Site
Mombelli P, Le Chapelain C, Munzinger N, Joliat E, Illarionov B, Schweizer W, Hirsch A, Fischer M, Bacher A, Diederich F (2013)
Exploiting specific interactions toward next-generation polymeric drug transporters
Wieczorek S, Krause E, Hackbarth S, Röder B, Hirsch A, Börner H (2013)
Journal of the American Chemical Society 135 (5): 1711-4DOI: 10.1021/ja311895z
2012
Total synthesis of (-)-doliculide, structure-activity relationship studies and its binding to F-actin
Matcha K, Madduri A, Roy S, Ziegler S, Waldmann H, Hirsch A, Minnaard A (2012)
ChemBioChem 13 (17): 2537-48DOI: 10.1002/cbic.201200512
The isoprenoid-precursor dependence of Plasmodium spp
van der Meer J, Hirsch A (2012)
Natural product reports 29 (7): 721-8DOI: 10.1039/c2np20013a
Metal-ion-induced shape switching: Stereoselective formation of a dinuclear Hg(II) double helicate from a hydrazonobis(acylhydrazone) ligand
Schaeffer G, Harrowfield J, Lehn J, Hirsch A (2012)
Biodynamers: self-organization-driven formation of doubly dynamic proteoids
Hirsch A, Buhler E, Lehn J (2012)
Journal of the American Chemical Society 134 (9): 4177-83DOI: 10.1021/ja2099134
Exploring the Ribose Sub-Pocket of the Substrate-Binding Site in Escherichia coli IspE: Structure-Based Design, Synthesis, and Biological Evaluation of Cytosines and Cytosine Analogues
Schütz A, Osawa S, Mathis J, Hirsch A, Bernet B, Illarionov B, Fischer M, Bacher A, Diederich F (2012)
2009
Bioconjugates to specifically render inhibitors water-soluble
Hirsch A, Diederich F, Antonietti M, Börner H (2009)
Soft Matter 6 (1): 88-91DOI: 10.1039/B915928B
2008
The Non-Mevalonate Pathway to Isoprenoid Biosynthesis: A Potential Source of New Drug Targets
Hirsch A, Diederich F (2008)
CHIMIA 62 (4): 226-230DOI: 10.2533/chimia.2008.226
Synthesis and characterization of cytidine derivatives that inhibit the kinase IspE of the non-mevalonate pathway for isoprenoid biosynthesis
Crane C, Hirsch A, Alphey M, Sgraja T, Lauw S, Illarionova V, Rohdich F, Eisenreich W, Hunter W, Bacher A, Diederich F (2008)
ChemMedChem 3 (1): 91-101DOI: 10.1002/cmdc.200700208
Inhibitors of the kinase IspE: structure-activity relationships and co-crystal structure analysis
Hirsch A, Alphey M, Lauw S, Seet M, Barandun L, Eisenreich W, Rohdich F, Hunter W, Bacher A, Diederich F (2008)
Organic & biomolecular chemistry 6 (15): 2719-30DOI: 10.1039/b804375b
2007
Phosphate recognition in structural biology
Hirsch A, Fischer F, Diederich F (2007)
Angewandte Chemie (International ed. in English) 46 (3): 338-52DOI: 10.1002/anie.200603420
Nonphosphate inhibitors of IspE protein, a kinase in the non-mevalonate pathway for isoprenoid biosynthesis and a potential target for antimalarial therapy
Hirsch A, Lauw S, Gersbach P, Schweizer W, Rohdich F, Eisenreich W, Bacher A, Diederich F (2007)
ChemMedChem 2 (6): 806-10DOI: 10.1002/cmdc.200700014
2006
Double conjugate addition of dithiols to propargylic carbonyl systems to generate protected 1,3-dicarbonyl compounds
Sneddon H, van den Heuvel A, Hirsch A, Booth R, Shaw D, Gaunt M, Ley S (2006)
The Journal of organic chemistry 71 (7): 2715-25DOI: 10.1021/jo052514s